PUBBLICAZIONI SCIENTIFICHE
RASSEGNA STAMPA E PUBBLICAZIONI SCIENTIFICHE
PCR (Polymerase chain reaction)
LABORATORIO DI METOLOGIE E TECNOLOGIE GENETICHE PCR (Polymerase chain reaction)
La PCR è un metodo di biologia molecolare, utile per amplificare (i.e. creare copie multiple di) DNA, inventato da Kary Mullis alla fine degli anni ’80. Lo scopo della PCR è di produrre un enorme numero di copie di una sequenza di DNA, sia essa un gene o parte di questo. Nella pratica corrente i principali componenti della PCR sono:
DNA stampo, che contiene il frammento da amplificare;
Due primers (Forward e Reverse), che determinano l’inizio e la fine della regione da amplificare;
DNA Polimerasi, che copia la regione da amplificare (si usano le DNA polimerasi di batteri termofili, che sono stabili ad elevate temperature; es. Taq Polimerasi);
Nucleotidi, per la sintesi di nuovo DNA;
Buffer, che fornisce l’ambiente chimico ideale per la DNA Polimerasi.
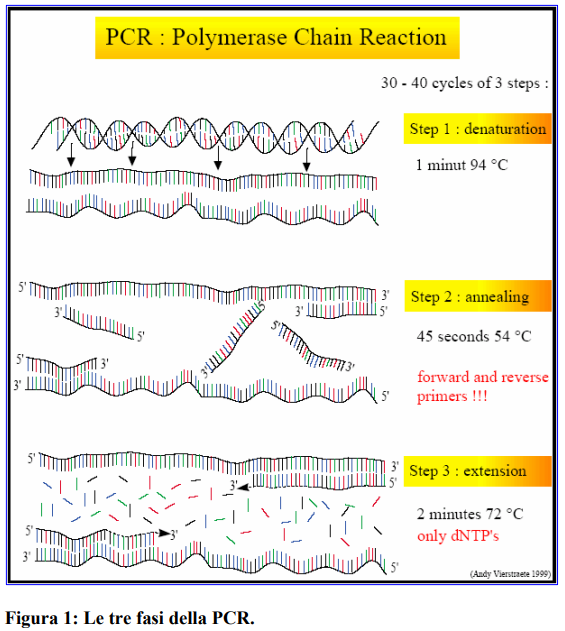
Il processo della PCR consta di tre steps principali che vengono ripetuti dalle 30 alle 40 volte, attraverso l’utilizzo di un termociclatore.
1). Denaturazione a 94o C (Melting): Durante la denaturazione, i due filamenti di DNA si separano e tutte le reazioni enzimatiche si arrestano (es. l’estensione del filamento del ciclo precedente);
2). Annealing a 50 – 60 o C: La temperatura di questo stadio dipende dai primers usati. Durante l’annealing vengono utilizzati due primers (Forward e Reverse), che si legano ai loro siti specifici. Si formano e si rompono continuamente i legami ionici tra i filamenti dei primers ed i singoli filamenti di DNA che funzionano da templato (filamento stampo). I primers che si appaiano esattamente con il filamento stampo formano i legami più stabili ed, in questo piccolo tratto di doppia elica, si lega la polimerasi, che così inizia a copiare il filamento stampo;
3). Estensione a 70 – 75 o C (Elongation): La temperatura di elongation dipende dalla DNA Polimerasi. Durante l’estensione, la Polimerasi estende i primers aggiungendo le basi (complementari al templato) all’estremità 3’. Il risultato sono 2 copie di DNA a doppio filamento.
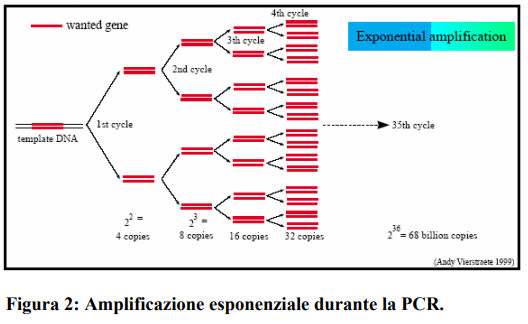
Poichè entrambi i filamenti sono copiati durante la PCR, si ha un aumento esponenziale del numero di copie della sequenza di DNA. X=X0(2N) N= NUMERO DI CICLI Con 100 molecole di DNA come stampo, dopo 30 cicli, la PCR produrrà PIU’ DI 100 MILIONI DI COPIE.
Caratteristiche della PCR
• Si possono utilizzare piccole quantità di DNA(ng);
• La qualità del DNA può anche non essere buona;
• Alta riproducibilità, specificità e sensibilità;
• Applicazioni cliniche e nella ricerca;
• Molto rapida e poco costosa;
• Puo’ essere usata solo se si conosce la sequenza del DNA.
A cosa serve la PCR?
La PCR può essere utilizzata in un’ampia varietà di analisi tra cui:
-Genetic fingerprinting (usata in medicina legale);
-Test di paternità; Studi di evoluzione e filogenesi;
-Identificazione di malattie ereditarie;
-Identificazione di malattie virali;
-Clonaggio di un gene;
-Analisi di DNA antico; Genotipizzazione di specifiche mutazioni e polimorfismi.
Epidemiologia di base
R. Beaglehole R. Bonita Department of Community Health and Department of Medicine University of Auckland Auckland New Zealand
T. KjeIlstrom Division of Environmental Health World Health Organization Geneva Switzerland Curatore responsabile dell’edizione italiana:
Gabriella Aggazzotti Cattedra di Metodologia epidemiologica e Igiene Facoltà di Medicina e Chirurgia Università degli Studi di Modena