Estrazione rapida dell’acido nucleico

LABORATORI CONDIVISI
- Nano Science and Technology (NSNT), Università di scienza e tecnologia di Hong Kong, Clear Water Bay, Kowloon, Hong Kong
- Shenzhen Shineway Hi-Tech Co., Ltd., Shenzhen 518112, Cina
- Dipartimento di Fisica, Università di Scienza e Tecnologia di Hong Kong, Clear Water Bay, Kowloon, Hong Kong
- Guangzhou HKUST Fok Ying Tung Research Institute, Nansha, Guangzhou 511458, Cina
- Dipartimento di Scienze dell’Oceano, Università di Scienza e Tecnologia di Hong Kong, Clear Water Bay, Kowloon, Hong Kong
- Materials Genome Institute, Università di Shanghai, Shanghai 200444, Cina
Microfluidica per cellule e altri organismi
Abstract
Il risultato della diagnostica e del rilevamento molecolari dipende in larga misura dalla qualità e dall’integrità dell’acido nucleico isolato. In questo lavoro, abbiamo sviluppato un dispositivo automatizzato di estrazione di acido nucleico miniaturizzato basato sul metodo delle microsfere magnetiche, costituito da quattro componenti tra cui un disco di elaborazione del campione e il relativo meccanismo di uscita di potenza rotante associato, un modulo di pipettaggio, un modulo magnete e un controller centrale esterno per consentire una preparazione del campione di acido nucleico personalizzabile e automatizzata. L’acido nucleico estratto usando cellule 293T è stato verificato utilizzando la reazione a catena della polimerasi in tempo reale (PCR) e i dati implicano un’efficienza comparabile a un processo manuale, con i vantaggi di eseguire un flessibile, risparmio di tempo (~ 10 min) e semplice preparazione del campione di acido nucleico.
Parole chiave: preparazione del campione ; acido nucleico ; DNA ; RNA
1. Introduzione
Con il rapido sviluppo della tecnologia della biologia molecolare negli ultimi anni, le tecnologie di diagnostica e rilevazione molecolare rappresentate dall’ibridazione dell’acido nucleico, dall’amplificazione dell’acido nucleico e dall’analisi della sequenza dell’acido nucleico sono diventate sempre più significative in molti campi. Tuttavia, la sfida fondamentale per tutte le moderne tecniche di rilevamento della biologia molecolare, come la reazione a catena della polimerasi (PCR), il sequenziamento ad alto rendimento, ecc. È come separare ed estrarre prontamente ed efficacemente l’acido nucleico genomico richiesto da campioni biologici complessi e diversificati, poiché la qualità e l’integrità (stato di degradazione) dell’acido nucleico isolato influiscono direttamente sui successivi risultati sperimentali [ 1]. Al momento, i ricercatori di tutto il mondo hanno fatto molti progressi nella tecnologia della separazione e dell’estrazione dell’acido nucleico.
Gli acidi nucleici sono ampiamente classificati in acido desossiribonucleico (DNA) e acido ribonucleico (RNA). Sin dalla sua prima scoperta nel 1869, molti ricercatori hanno compiuto sforzi incessanti nell’estrazione di acidi nucleici e hanno migliorato vari materiali e reagenti per l’estrazione di acido nucleico. I risultati della ricerca sulle pietre miliari includono: tecnica di estrazione con fenolo [ 2 ], metodo con dodecil solfato di sodio (SDS) [ 3 ] e tecnica di estrazione con fenolcoroformio acido guanidinio (AGPC) [ 4 , 5]. Numerose famose società di reagenti biologici hanno sviluppato vari kit di estrazione di acido nucleico basati su questi metodi di estrazione di acido nucleico convenzionali per la separazione e l’estrazione di DNA e RNA da un’ampia varietà di campioni di tessuto. I metodi convenzionali di estrazione dell’acido nucleico spesso includono precipitazione e centrifugazione, che richiedono un ampio numero di passaggi e quindi sono complicati, richiedono molto tempo (richiedono fino a 3 ore e molto più a lungo se incubati durante la notte) [ 6] e difficile ottenere l’automazione miniaturizzata. La maggior parte dei metodi richiede che gli operatori siano a diretto contatto con reagenti chimici tossici. Pertanto, con il rapido sviluppo della biologia molecolare e della scienza dei materiali polimerici, il metodo convenzionale di separazione ed estrazione degli acidi nucleici dai sistemi in fase liquida è stato gradualmente sostituito da nuovi metodi basati su portatori di adsorbato in fase solida [ 7 , 8 ]. Tali metodi emergenti di separazione ed estrazione dell’acido nucleico comprendono principalmente: metodo delle particelle di vetro [ 9 ], metodo delle matrici di silice [ 10 , 11 ], metodo di scambio anionico [ 12 ] e metodo di estrazione basato su microsfere magnetiche [ 13]. Indipendentemente dal metodo utilizzato per separare ed estrarre acidi nucleici, in generale, le fasi operative di tali metodi possono essere principalmente suddivise in quattro parti [ 7 , 8 , 14 ]. La prima parte consiste nell’utilizzare la lisi per promuovere la distruzione cellulare e liberare gli acidi nucleici. La seconda parte consiste nell’adsorbire specificamente gli acidi nucleici rilasciati su uno specifico trasportatore, con questo specifico trasportatore ha una forte affinità e adsorbimento solo per gli acidi nucleici, ma non ha affinità per altri componenti biochimici come proteine, polisaccaridi e lipidi. La terza parte è lavare con un tampone di lavaggio specifico per rimuovere le impurità dell’acido non nucleico e l’ultima parte è eluire l’acido nucleico adsorbito sul veicolo specificato per ottenere l’acido nucleico purificato [ 15].
L’estrazione di acidi nucleici con il metodo basato sulla colonna di spin è stata ampiamente utilizzata e la maggior parte dei kit di estrazione del DNA plasmidico sul mercato sono stati sviluppati in base al metodo basato sulla colonna di spin. Il metodo adotta uno speciale materiale di adsorbimento con matrice di silicio, che è caratterizzato come segue: in presenza di un tampone di acido cloridrico elevato, il DNA può essere specificamente assorbito, le impurità possono essere rimosse con una serie di fasi di lavaggio e il basso contenuto di sale il tampone alcalino può eluire il DNA legato alla colonna di adsorbimento [ 14]. Tuttavia, lo svantaggio di questo metodo è che il campione richiesto è grande, consumando quindi molti campioni. Inoltre, l’applicazione di questo metodo su alcuni campioni rari è notevolmente limitata. Allo stesso tempo, il metodo della colonna di spin richiede una ripetuta centrifugazione durante il processo, che non è adatto per un funzionamento automatizzato ad alto rendimento. Soprattutto nel campo della diagnosi genetica, del monitoraggio e del controllo di focolai improvvisi, l’uso del metodo basato sulla colonna di spin per estrarre acidi nucleici richiede un gran numero di operatori e attrezzature per soddisfare la domanda. Dagli anni ’90, a causa di varie carenze nel metodo della colonna di spin, al fine di adattarsi ai requisiti di elevata produttività, alta sensibilità e funzionamento automatizzato dei moderni esperimenti di test di biologia molecolare,16]. Questo metodo è la combinazione perfetta di nanotecnologia e biotecnologia poiché le microsfere magnetiche sono microsfere magnetiche composite ad alta affinità (in genere da 1 a 100 nm) formate combinando una particella magnetica inorganica con un materiale polimerico. Questo metodo è vantaggioso per altri metodi di estrazione di acido nucleico, che si riflettono principalmente in: (1) Può realizzare operazioni e automazione ad alto rendimento; (2) L’operazione è semplice e fa risparmiare tempo, l’intero processo di estrazione prevede solo quattro passaggi e l’intero processo può essere completato in 40 minuti; (3) è sicuro e non tossico; (4) Il legame specifico di sfere magnetiche e acido nucleico rende l’acido nucleico estratto elevato in purezza e concentrazione; (5) Basso costo e può essere applicato in un ampio numero di applicazioni.17 , 18 ]. Un grande miglioramento nell’estrazione dell’acido nucleico da parte di microsfere magnetiche è l’uso di una microsfera magnetica composita ad alta affinità, formata dalla combinazione di particelle magnetiche inorganiche e materiali polimerici. A causa delle sue numerose proprietà di microsfere polimeriche e particelle magnetiche, è uniformemente e stabilmente disperso nella soluzione senza un campo magnetico esterno e può essere facilmente e rapidamente separato una volta aggiunto il campo magnetico esterno.
La tecnologia microfluidica è nata dal concetto di un sistema microelettromeccanico basato sulla tecnologia MEMS, come proposto da Manz et al. nei primi anni ’90 [ 19 ]. Lo scopo di questo sistema è di trasferire le funzioni del laboratorio su dispositivi portatili e persino su chip, attraverso la miniaturizzazione e l’integrazione dei sistemi di analisi chimica. Il cuore di questa tecnologia sono i chip microfluidici, in cui una serie di microcanali è fabbricata su un piccolo chip. Attraverso la manipolazione e il controllo dei microfluidi nel microcanale, vengono realizzate tutte le funzioni di laboratorio chimico e biologico. Diverse tecniche di estrazione di acido nucleico con chip microfluidico sono state segnalate per isolare le cellule bersaglio dai campioni di sangue usando filtri di tipo a stramazzo [ 20 ], filtri per pori [21 ], o sfere immunomagnetiche specifiche del patogeno [ 22 ]. Le cellule bersaglio catturate vengono introdotte in una camera di reazione PCR su un chip e il DNA viene rilasciato dalla lisi termica delle cellule. Tuttavia, poiché la miscela presente nei detriti cellulari può inibire il processo di PCR, la maggior parte dei chip microfluidici segnalati per l’estrazione del DNA richiedono fasi preliminari di elaborazione del campione fuori chip. Per l’estrazione del DNA sul chip, il metodo più comune attualmente utilizzato è quello di estrarre il DNA dai lisati cellulari usando microsfere magnetiche rivestite con silice o gruppi funzionali (carbossi [ 22 ], ammina [ 23 ], biotina [ 24 ], sonde nucleotidiche [ 25 ] ). Inoltre, sono stati condotti studi di successo sull’intrappolamento dielettroforetico [ 26] e isotachoforesi [ 27 ] su chip microfluidici di purificazione del DNA.
Sebbene la maggior parte degli sviluppi nel campo della diagnostica molecolare si sia concentrata sul miglioramento dei metodi per rilevare e identificare gli analiti target correlati alla malattia, meno attenzione è stata dedicata allo sviluppo di sistemi per la purificazione dei campioni. Il nostro obiettivo è sviluppare un sistema personalizzabile, automatizzato e miniaturizzato per la preparazione del campione di acido nucleico basato sul metodo delle microsfere magnetiche. In questo lavoro, abbiamo sviluppato un dispositivo automatizzato di estrazione di acido nucleico miniaturizzato costituito da quattro componenti tra cui un disco di elaborazione del campione e il relativo meccanismo di uscita di potenza rotante associato, un modulo di pipettaggio, un modulo magnete e un controller centrale esterno. Il dispositivo è stato progettato in modo discreto per garantire la sua portabilità (dimensioni 220 × 165 × 210 mm e 3 kg di peso), efficienza e facilità d’uso. La nostra piattaforma integra le funzioni di lisi, legame, lavaggio ed eluizione e possono essere personalizzati in base a diversi protocolli di estrazione per soddisfare le diverse esigenze degli utenti. Abbiamo analizzato le prestazioni del dispositivo utilizzando cellule 293T, gli acidi nucleici estratti sono stati rilevati mediante PCR in tempo reale e verificati mediante elettroforesi. Il processo manuale di estrazione dell’acido nucleico richiede in genere da 40 a 50 min e l’estrazione del DNA basata su microchip precedentemente riportata costa 12 min [28 ] a 15 min [ 22 ], il nostro dispositivo ha ulteriormente ridotto la durata del processo entro 10 min. Gli strumenti di estrazione di acido nucleico automatizzati commercializzati (ad es. QIAcube di QIAGEN, Hilden, Germania) e robot o piattaforme per la manipolazione di liquidi (ad es. QIAgility di QIAGEN) hanno funzioni simili al nostro dispositivo ma con dimensioni molto maggiori e un peso maggiore (QIAcube è 71,5 kg e QIAgility è 41 kg). Le dimensioni ridotte, la leggerezza e il rapido tempo di elaborazione rendono il nostro dispositivo adatto per l’uso futuro nei test presso i centri di cura.
2. Materiali e metodi
2.1. Idea di design
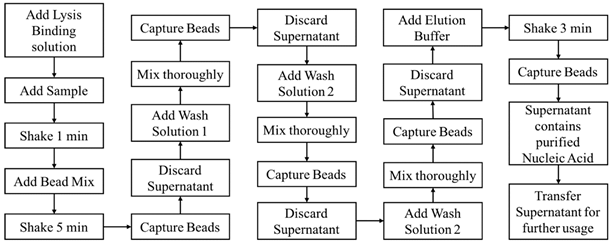
Rispetto al metodo convenzionale di estrazione dell’acido nucleico, l’estrazione di microsfere magnetiche e la purificazione dell’acido nucleico presentano vantaggi incomparabili. Il processo dell’esperimento è semplice, facile da usare e può far risparmiare tempo poiché le fasi di centrifugazione e precipitazione che richiedono tempo nei metodi convenzionali non sono necessarie quando si utilizzano particelle magnetiche. Inoltre, questo sistema può essere utilizzato su processi sia manuali che automatizzati. Il processo è anche sicuro e non tossico, poiché non utilizza i reagenti tossici come benzene e cloroformio richiesti nei metodi convenzionali. Il legame specifico di particelle magnetiche e acido nucleico rende l’acido nucleico estratto alto in purezza e concentrazione. L’estrazione dell’acido nucleico del tallone magnetico può generalmente essere suddivisa in quattro fasi: lisi, legame, lavaggio ed eluizione.Figura 1 , abbiamo progettato e realizzato un dispositivo miniaturizzato automatizzato per la realizzazione dell’automazione di tutti i passaggi e le funzioni, riducendo in tal modo la forza lavoro e migliorando l’efficienza di purificazione.
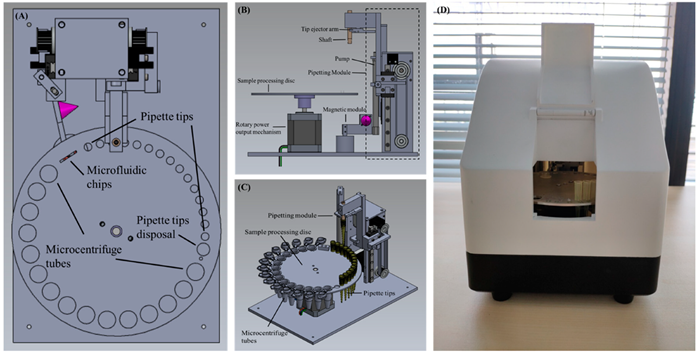
2.2. Configurazione e meccanismo del dispositivo
Il dispositivo è costituito da un disco di elaborazione del campione e dal relativo meccanismo di uscita di potenza rotante associato, un modulo di pipettaggio, un modulo magnetico e un controller centrale esterno per controllare i comandi di esecuzione di tutti i moduli in parallelo o in parallelo. Lo schema del dispositivo è mostrato in Figura 1 . Il disco di elaborazione del campione, come mostrato nella Figura 2A, ha numerosi fori rotondi di diverse dimensioni sulla circonferenza più esterna. In particolare, i fori con le dimensioni più piccole servono per il posizionamento della punta della pipetta, i fori con la dimensione più grande servono per il posizionamento di provette da microcentrifuga da 1,5 ml e vi è un foro di medie dimensioni con una circonferenza leggermente superiore alla circonferenza massima della punta della pipetta, che viene utilizzato per lo smaltimento di puntali di pipette usati e rifiuti di reagente. Il foro rettangolare serve per posizionare il chip microfluidico, che viene utilizzato per ulteriori PCR. Il meccanismo di uscita di potenza rotante si trova sotto il disco di elaborazione del campione ed è collegato al suo centro per regolare l’angolo di rotazione del disco, come mostrato nella Figura 2B. Il modulo di pipettaggio comprende un albero per contenere i puntali delle pipette, un braccio di espulsione dei puntali per la rimozione dei puntali e una pompa utilizzata per aspirare o distribuire il liquido dal puntale della pipetta monouso. Inoltre, due motori passo-passo si collegano ciascuno a una cinghia di scorrimento ( Figura 2 C) sono accoppiati al controller centrale esterno separatamente per guidare l’albero e la pompa per spostarsi verticalmente rispetto al piano orizzontale. Il modulo magnete include un magnete a cono ( Figura 2A, B) colorato in viola) per fornire un campo magnetico esterno e un servomotore si collega al controller centrale esterno per dominare la presenza e l’assenza del campo magnetico. Inoltre, il disco di elaborazione del campione, il meccanismo di uscita di potenza rotante, il modulo di pipettaggio e il modulo magnete sono integrati in una scatola portatile con una dimensione di 220 × 165 × 210 mm, come mostrato nella Figura 2 D, e l’intero dispositivo ha un peso di circa 3 kg. All’interno della scatola portatile, una luce UV controllata dal controller esterno si trova sulla parte superiore per esporre e decontaminare il dispositivo, eliminando quindi la contaminazione tra le corse.
2.3. Processo di esperimento
Come descritto nella sezione precedente, il disco di elaborazione del campione ha varie dimensioni di fori rotondi situati sulla circonferenza più esterna. A scopo sperimentale, abbiamo assegnato etichette per ciascun foro in base alle loro dimensioni, come mostrato nella Figura 3 A. I fori più piccoli per posizionare le punte delle pipette erano etichettati S, il foro di dimensioni medie per lo smaltimento dei rifiuti era etichettato M e i fori più grandi per il posizionamento Le provette per microcentrifuga sono state etichettate con L. Prima di iniziare l’esperimento, dovevamo aggiungere ciascun reagente con un volume specificato alle provette per microcentrifuga e quindi posizionarle sui fori con etichetta prestabiliti, come mostrato nella configurazione iniziale della Figura 3B. Il campione di cellule che abbiamo usato erano 293 cellule T coltivate nel terreno di Eagle modificato di Dulbecco (DMEM; Gibco, Carlsbad, CA, USA) con siero bovino fetale al 10% (FBS; Gibco, USA) e 1% di penicillina / streptomicina a 37 ° C in 5% di CO 2 con un importo iniziale di 2 × 10 6le cellule. Il programma software nel controller centrale esterno, che controlla ogni modulo in modo indipendente, era stato preimpostato secondo il protocollo di questo esperimento, e il programma poteva essere comodamente personalizzato se si utilizzavano protocolli o kit diversi. Dopo aver posizionato tutti i puntali di pipetta e le provette per microcentrifuga necessari sui fori corrispondenti, è possibile avviare il processo di preparazione del campione semplicemente premendo il pulsante di avvio sul controller centrale esterno. L’intero processo potrebbe quindi essere condotto automaticamente dal programma preimpostato. I processi di dettaglio eseguiti dal dispositivo sono descritti di seguito e il flusso di processo è mostrato nella Figura 3B. In primo luogo, il disco di elaborazione del campione ruotava in modo che S1 fosse proprio sotto l’albero, l’albero si spostava verticalmente verso il basso per prendere una punta della pipetta e si spostava verso l’alto dopo. Per trasferire il reagente da L2 a L1, il disco di elaborazione del campione ruotava in modo che L2 fosse sotto l’albero, l’albero si spostava verso il basso in una posizione in cui la punta della pipetta veniva unita nel reagente e la pompa si spostava verso l’alto per aspirare il reagente nella punta della pipetta . Il disco di elaborazione del campione quindi ruotava su L1, l’albero e la pompa si spostavano verso il basso per erogare il reagente, la pompa si spostava su e giù ripetutamente per aspirare e distribuire il liquido per miscelare accuratamente i reagenti. La punta della pipetta usata deve essere sostituita prima del passaggio successivo, quindi il disco di elaborazione del campione ruotato in modo che M1 fosse sotto l’albero, e l’albero si spostava verso il basso per innescare il braccio di espulsione della punta per smaltire la punta della pipetta usata e un contenitore per rifiuti era posizionato sotto per conservare le punte usate e i rifiuti di reagente. Dopo l’introduzione di un nuovo puntale da S2, il liquido in L4 è stato trasferito in L3 e miscelato accuratamente. Successivamente, la punta usata è stata espulsa attraverso M1, una nuova punta è stata prelevata da S3 e il liquido è stato trasferito da L1 a L9, quindi da L3 a L9 e miscelato accuratamente. Il magnete è stato quindi spostato su L9 per applicare il campo magnetico al fine di catturare magneticamente le sfere di legame, con il campo magnetico attivato, l’albero spostato verso il basso per aspirare il surnatante, quindi ha scartato la punta usata insieme al surnatante nel contenitore dei rifiuti attraverso M1 . Un nuovo suggerimento è stato preso da S4, e il liquido è stato trasferito da L5 a L9 e miscelato accuratamente. Seguendo questo, il campo magnetico fu applicato a L9 e il surnatante fu aspirato e disposto. È stato preso un nuovo suggerimento da S5 e il liquido è stato trasferito da L6 a L9 e i passaggi precedenti sono stati ripetuti. Successivamente, è stato prelevato un nuovo puntale da S6 e il liquido è stato trasferito da L7 a L9 con il processo sopra descritto ripetuto nuovamente. Fino a questo punto del processo, tutte le fasi di aggiunta, miscelazione e lavaggio dei reagenti richieste in questo protocollo erano state completate dal sistema. Infine, un nuovo suggerimento è stato preso da S7 e il buffer di eluizione situato da L8 a L9 è stato trasferito e miscelato accuratamente. La punta usata è stata quindi espulsa attraverso M1 e una nuova punta è stata prelevata da S8, un campo magnetico è stato applicato a L9 e il surnatante è stato aspirato, l’RNA purificato era anche nel surnatante per ulteriori applicazioni a valle. Durante questo esperimento, abbiamo dispensato il surnatante, che conteneva RNA purificato in una provetta da microcentrifuga vuota posizionata su L10 per ulteriori esperimenti di trascrizione inversa dell’RNA e di verifica della PCR. Inoltre, il campione acquisito potrebbe essere erogato direttamente nel chip microfluidico posizionato sul disco di elaborazione del campione per esperimenti a valle in futuro.
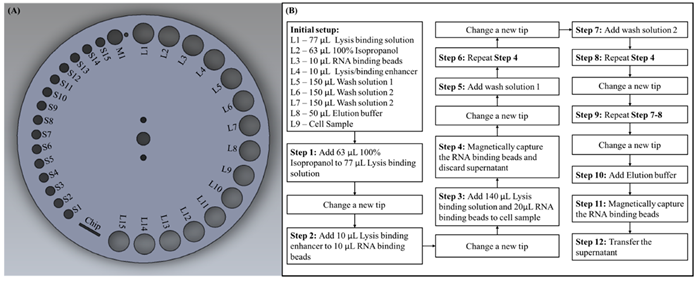
Tuttavia, il flusso di processo mostrato in Figura 3B è progettato in conformità con i reagenti e il protocollo che abbiamo scelto nel nostro esperimento. Si può facilmente vedere che avevamo occupato da L1 a L9 e da S1 a S8, rimanevano ancora molti fori di posizionamento da utilizzare se il protocollo richiedeva più reagenti. Poiché ogni fase dell’esperimento consisteva in molte micro fasi preprogrammate nel controller esterno, l’intero processo dell’esperimento poteva essere facilmente personalizzato per soddisfare le diverse esigenze. Ad esempio, al punto 3, per aggiungere 140 μL di soluzione legante di lisi in L1 al campione di cella L9, il disco di elaborazione del campione è stato programmato per ruotare in modo che L1 fosse sotto l’albero, l’albero quindi spostato verticalmente verso il basso fino a quando la punta della pipetta era unito nel reagente. Successivamente, la pompa si è spostata verso l’alto per aspirare il liquido nella pipetta, e l’albero si è spostato indietro in modo che il disco di elaborazione del campione potesse essere ruotato fino a quando L9 era sotto l’albero. Infine, l’albero si è mosso verso il basso e così ha fatto la pompa per erogare il reagente in L9. Il grado di rotazione del disco di elaborazione del campione, la distanza mobile dell’albero e la pompa sono stati tutti controllati dal controller centrale esterno inserendo un valore preimpostato. Pertanto, se l’utente desidera trasferire il reagente da L10 a L11, l’utente può semplicemente programmarlo nel controller centrale esterno. Se l’utente ha solo bisogno di due fasi di lavaggio, può facilmente eliminare il programma relativo al passaggio 9 (in e la pompa erano tutte controllate dal controller centrale esterno inserendo un valore preimpostato. Pertanto, se l’utente desidera trasferire il reagente da L10 a L11, l’utente può semplicemente programmarlo nel controller centrale esterno. Se l’utente ha solo bisogno di due fasi di lavaggio, può facilmente eliminare il programma relativo al passaggio 9 (in e la pompa erano tutte controllate dal controller centrale esterno inserendo un valore preimpostato. Pertanto, se l’utente desidera trasferire il reagente da L10 a L11, l’utente può semplicemente programmarlo nel controller centrale esterno. Se l’utente ha solo bisogno di due fasi di lavaggio, può facilmente eliminare il programma relativo al passaggio 9 (inFigura 3 B) nel controller programmabile.
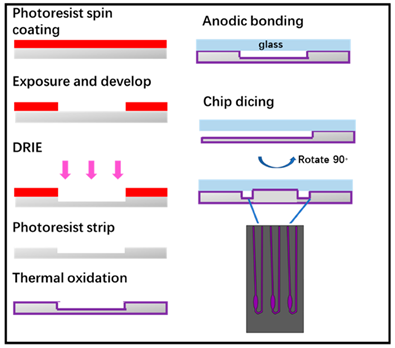
2.4. Fabbricazione di microchip
Il microchip è stato fabbricato con la tecnologia litografica standard, come mostrato nella Figura 4 . Successivamente, i wafer di silicio lucido a doppia faccia da 4 pollici sono stati puliti in soluzione di piranha e soluzione di acido fluoridrico. Il substrato a motivi fotolitografici per primo è stato sottoposto al processo di discesa in O 2plasma. Quindi, l’attacco ionico indotto di reazione al plasma accoppiato (ICP-DRIE) è stato applicato per incidere il silicio per formare pozzi e canali per eseguire reazioni in tempo reale di PCR (RT-PCR). Dopo che il fotoresist è stato rimosso dal plasma, il wafer ha sviluppato ossido termico con uno spessore di 1000 Å come strato di passivazione usato per evitare l’adsorbimento non specifico dei componenti della PCR. Il wafer di ossido è stato legato con un wafer di vetro da 4 pollici mediante legame anodico per formare una camera di PCR chiusa e infine il wafer è stato tagliato in singoli microchip. Vale la pena notare che abbiamo progettato l’ingresso e l’uscita del chip sul lato del chip anziché la sua parte superiore e la porta di ingresso e uscita potrebbe essere aperta subito dopo il taglio del wafer. Questo design potrebbe evitare i processi di perforazione o punzonatura della porta di ingresso e uscita, oltre a rendere più semplice per il nostro dispositivo l’aggiunta di acido nucleico estratto nel microchip. Il microchip progettato per questo esperimento ha tre camere di reazione ciascuna con un volume di reazione di 2 μL.
2.5. Trascrizione inversa dell’RNA
L’RNA totale è stato trascritto in modo inverso nel cDNA con il seguente protocollo: la miscela di RNA conteneva 2 μL di modello (estratto usando il dispositivo miniaturizzato automatizzato come descritto sopra), 1 μL di primer (Thermal Fisher Scientific, Carlsbad, CA, USA), 1 μL di dNTP Thermal Fisher Scientific, USA) e 6 μL ddH 2 O per portare il volume a 10 μL. La miscela è stata quindi riscaldata a 65 ° C per 5 minuti e il campione è stato rapidamente raffreddato su ghiaccio per 2 minuti. Una miscela di reazione è stata preparata in una nuova provetta separata contenente 4 μL 5 × tampone (Thermal Fisher Scientific, USA), 0,5 μL di enzima RT (Thermal Fisher Scientific, Stati Uniti), 0,5 μL di inibitore (Thermal Fisher Scientific, Stati Uniti) e 5 μL ddH 2O per formare una soluzione da 10 μL. Successivamente, 10 μL di miscela di reazione sono stati aggiunti a 10 μL di miscela di RNA, quindi il campione è stato incubato a 42 ° C per 60 minuti seguito da 25 ° C per 5 minuti.
2.6. Rilevamento dell’espressione genica mediante PCR in tempo reale
Il cDNA è stato amplificato dalla PCR in tempo reale e i geni target di amplificazione Actin e GAPDH sono stati verificati mediante elettroforesi su gel di agarosio al 2%. I primer GAPDH sono costituiti da un primer forward CAT GAG AAG TAT GAC AAC GCC T e un primer reverse AGT CCT TCC ACG ATA CCA AAG T, che producono un prodotto PCR con una dimensione di 113 bp. I primer Actin sono stati primer forward GAG CAC AGA GCC TCG CCT TT e primer reverse TCA TCA TCC ATG GTG AGC TGG C hanno prodotto un prodotto PCR con una dimensione di 70 bp. Ogni reazione RT-PCR è stata eseguita in 10 μL 2 × super mix Bio-Rad, 1 μL di reazione RT come cDNA, primer 0,5 μL (10 μM) e 8,5 μL ddH 2O. I parametri ciclici hanno comportato la denaturazione a 95 ° C per 3 minuti, seguita da 40 cicli di 95 ° C per 30 se 60 ° C per 1 minuto. Ogni espressione genica bersaglio è stata amplificata da due esperimenti ripetuti. La RT-PCR è stata eseguita utilizzando LineGene 9600 Plus (Bioer) e il suo valore di soglia del ciclo (Ct) è stato determinato.
2.7. materiale
Le cellule 293T sono state coltivate in DMEM (DMEM; Gibco, USA) con 10% FBS (FBS; Gibco, USA) e 1% di penicillina / streptomicina a 37 ° C in 5% di CO 2 , MagMAX TM -96 Total RNA Isolation Kit (Thermal Fisher Scientific, Carlsbad, CA, USA), 100% isopropanolo, 100% etanolo, kit di sintesi cDNA Max Strand H Minus First Strand (Thermal Fisher Scientific, USA), iQ TM SYBR Green Supermix (BIO-RAD, Foster City, CA, USA ), NanoDrop TM One (Thermal Fisher Scientific, Stati Uniti).
3. Risultati e discussioni
Al fine di testare le prestazioni del dispositivo miniaturizzato automatizzato per la preparazione del campione di acido nucleico, abbiamo condotto trascrizione inversa e PCR in tempo reale utilizzando il campione ottenuto dal dispositivo. I risultati sono mostrati in Figura 5 . Prima del test, l’RNA totale è stato estratto da cellule 293T mediante un processo completamente automatizzato utilizzando il nostro dispositivo automatizzato di preparazione del campione di acido nucleico miniaturizzato. La concentrazione di RNA totale è stata quantificata da NanoDrop TMUno ed è stato trovato per essere 500 ng / μL. L’RNA totale è stato quindi trascritto in modo inverso nel cDNA e abbiamo eseguito la PCR in tempo reale utilizzando il protocollo come descritto in Materiali e metodi. Poiché nella trascrizione sono stati utilizzati 2 μL di RNA totale, la concentrazione di cDNA è di 50 ng / μL, quindi aggiungere 1 μL di cDNA in ciascuna reazione di PCR equivale all’aggiunta di 50 ng di campione in ciascuna reazione di PCR. Abbiamo quindi usato l’espressione genica di Actin e GAPDH come standard nell’amplificazione della PCR. Figura 5A mostra i risultati dell’amplificazione PCR in tempo reale sullo strumento commerciale PCR in tempo reale (BIOER) dell’espressione genica di Actin e GAPDH. La curva superiore nera e la luce blu rappresentano due risultati ripetuti di amplificazione RT-PCR di Actin, e la curva inferiore blu e rossa rappresentano due risultati ripetuti di amplificazione RT-PCR della gliceraldeide 3-fosfato deidrogenasi (GAPDH). Il valore Ct di Actin e GAPDH era rispettivamente di 14 e 16, a dimostrazione del fatto che l’RNA veniva estratto con successo dalla cellula campione usando un processo completamente automatizzato senza intervento umano. Il valore Ct mostra anche una concentrazione piuttosto elevata del prodotto estratto.
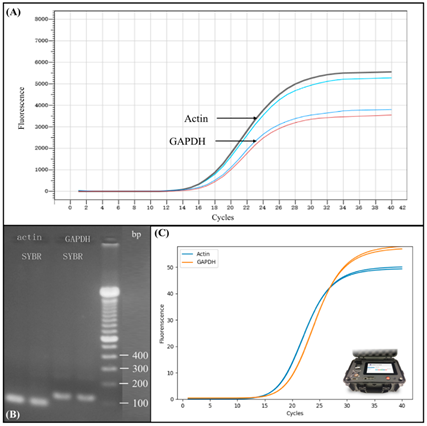
Figura 5. Verifica dei risultati della reazione a catena della polimerasi (PCR) in tempo reale utilizzando l’RNA totale estratto da cellule 293T utilizzando un dispositivo automatizzato di preparazione del campione di acido nucleico miniaturizzato. ( A ) Risultati dell’amplificazione della PCR in tempo reale sullo strumento commerciale di PCR in tempo reale (BIOER) dell’espressione genica di Actin e GAPDH. ( B ) Separazione del gel di elettroforesi e rilevazione dell’espressione genica di Actina e GAPDH come verifica del risultato dell’amplificazione riuscita. ( C ) Risultati di amplificazione PCR comparabili in tempo reale sullo strumento PCR in tempo reale microchip portatile che utilizza lo stesso campione e protocollo.
Per verificare l’RNA estratto dalle cellule 293T, abbiamo condotto un’analisi a valle utilizzando la separazione gel elettroforesi convenzionale. La Figura 5 B mostra i risultati dell’elettroforesi dell’espressione genica di Actina e GAPDH come verifica del risultato di amplificazione. Il marker che abbiamo usato, come mostrato nella riga all’estrema destra nella Figura 5 B, era la scala del DNA Thermo Fisher Scientific da 100 bp. I risultati mostrano che l’RNA estratto tramite post-PCR dal dispositivo automatizzato di preparazione dei campioni potrebbe essere separato con successo e chiaramente rilevato usando l’elettroforesi su gel convenzionale, indicando la fattibilità del nostro dispositivo per un’efficace preparazione del campione di acido nucleico.
Al fine di verificare ulteriormente le prestazioni del nostro dispositivo sviluppato, abbiamo confrontato i risultati dell’amplificazione PCR in tempo reale da strumenti PCR in tempo reale di grandi dimensioni commercializzati con il dispositivo PCR in tempo reale microchip portatile, come mostrato nella Figura 5 C. La curva di amplificazione mostrata in La Figura 5 C è stata ottenuta utilizzando lo stesso RNA totale estratto dal nostro dispositivo miniaturizzato automatizzato seguito dalla trascrizione inversa nel cDNA, come precedentemente descritto. Tuttavia, il DNA è stato amplificato utilizzando un altro dei nostri dispositivi, il dispositivo per PCR in tempo reale con microchip portatile, come mostrato nella Figura 5C, piuttosto che uno strumento PCR commercializzato. Ogni reazione di PCR è stata effettuata usando 2 μL di miscela PCR, come precedentemente descritto. Il valore Ct di Actin e GAPDH era rispettivamente di 14 e 16, il che era identico ai risultati dell’esperimento ottenuti da grandi strumenti di PCR commerciali. Questi risultati coerenti dimostrano che l’RNA totale estratto aveva un’elevata purezza, poiché anche un volume di 2 microlitri di miscela PCR poteva dimostrare lo stesso risultato, dimostrando così l’efficienza del nostro dispositivo miniaturizzato automatizzato. Inoltre, il nostro strumento PCR portatile in tempo reale con microchip ha generato lo stesso valore Ct degli strumenti PCR commercializzati.
4. Conclusioni
In conclusione, abbiamo sviluppato un dispositivo miniaturizzato automatizzato per ottenere l’automazione della purificazione dell’acido nucleico da campioni reali. Questo dispositivo può adattarsi a diversi protocolli di estrazione configurando il software nel controller centrale esterno per soddisfare esigenze personalizzate e il campione di acido nucleico estratto potrebbe essere introdotto direttamente nel microchip per ulteriori applicazioni a valle. Questo metodo consente un’estrazione di acido nucleico facile e che fa risparmiare tempo, indipendentemente dall’esperienza dell’operatore. I dati hanno indicato che l’estrazione di RNA totale automatizzata era equivalente alle prestazioni usando un processo manuale, ma l’estrazione automatizzata fa risparmiare più tempo con l’intero processo ridotto a 10 min. L’RNA totale estratto da cellule 293T può essere verificato mediante PCR con elettroforesi post gel o qPCR. Il processo non richiede strumenti esterni per la centrifugazione o la precipitazione ne garantisce la portabilità. In futuro, saremo in grado di integrare il dispositivo miniaturizzato automatizzato con il nostro strumento PCR in tempo reale microchip portatile per ottenere un processo sperimentale completamente automatizzato per applicazioni biomediche pratiche.
Contributi dell’autore
Concettualizzazione, RT e YG; software, RT; validazione, LZ, CH e XC; risorse, XT e YC; cura dei dati, QS e KL; scrittura: preparazione di bozze originali, RT; scrittura: revisione e modifica, RT; supervisione, WW; acquisizione di finanziamenti, WW e XG
finanziamento
Questa ricerca è stata finanziata da “Hong Kong RGC, Grant number 16219216”, “Shanghai Pujiang Program (17PJ1402800)” e “National Natural Science Foundation of China (21775101)”.
Ringraziamenti
Gli autori desiderano riconoscere il supporto dei tecnici e dei ricercatori del Design and Manufacturing Services Facility (DMSF), Nanosystem Fabrication Facility (NFF) presso l’Università di Scienza e Tecnologia di Hong Kong e Shenzhen Shineway Hi-tech Co., Ltd.
